基因密码子扩展技术(或称非天然氨基酸插入技术)通过在蛋白质特定位点插入人造的非天然氨基酸,实现对蛋白质结构和功能的精确调控,已被广泛应用于蛋白质功能研究和药物工程等领域。该技术利用工程化的氨酰tRNA合成酶连接非天然氨基酸(UAA)和相应的工程化tRNA,介入目标蛋白质的翻译过程,将原本不编码氨基酸的无义密码子编码为非天然氨基酸。
2022年,针对内源基因发生无义突变导致蛋白生成异常的遗传性疾病(如假性肥大型肌营养不良DMD,囊性纤维化CF,粘多糖贮积症I-H型MPS I-H等),夏青团队基于蛋白质翻译终止机制的研究,利用基因密码子扩展技术中的重要元件,即工程化“tRNA-aaRS”正交系统恢复模型小鼠中致病基因的表达。该研究验证了基因密码子扩展系统能够在模型动物体内稳定表达,并可以精准通读内源性的无义突变位点,恢复致病蛋白的全长表达,起到缓解疾病症状的疗效,既提出了一种通用型无义突变疾病治疗的新方法,也是基因密码子扩展技术用于遗传性疾病在体治疗的大胆尝试(Nat Biomed Eng, 2022.2),这是首次将这项技术应用于 DMD 治疗中。

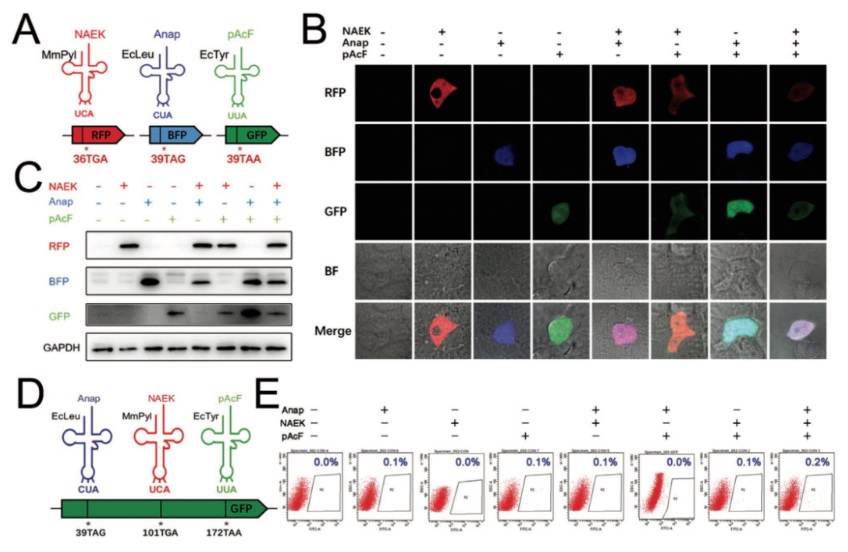
非天然氨基酸可位点特异性地引入目标蛋白,且对蛋白结构的扰动较小,通过更换氨基酸的侧链基团更可实现修饰蛋白的功能多样化,在细菌、哺乳动物细胞等的蛋白质修饰中表现优秀,拥有广阔的发展空间。但在哺乳动物细胞中联用多套非天然氨基酸系统,对研究人员来说仍是个艰巨的挑战。为了更好地解决这个问题,夏青团队从通读效率和正交性两方面选择了三种非天然氨基酸掺入系统的最优组合,通过对 MmPylRS/tRNAMmPyl、EcTyrRS/tRNAEcTyr和EcLeuRS/tRNAEcLeu 的优化,使其分别解码哺乳动物细胞中的三种无义密码子,也即 UGA、UAA 和 UAG,并编码不同功能的非天然氨基酸。2022 年 7 月,相关论文以《优化 eRF1 以实现哺乳动物细胞中三种非天然氨基酸的遗传编码》(Optimizing eRF1 to enable the genetic encoding of three distinct Non-canonical amino acids in mammalian cells)为题在Advanced Biology上发表。该研究首次实现在哺乳动物细胞中,将三个不同的非天然氨基酸位点特异性地整合到一个蛋白质中,建立了在真核体系中进行多位点特异性蛋白修饰的新工具,突破了合成生物学领域的一道壁垒,为无义突变罕见病的治疗提供新的策略。

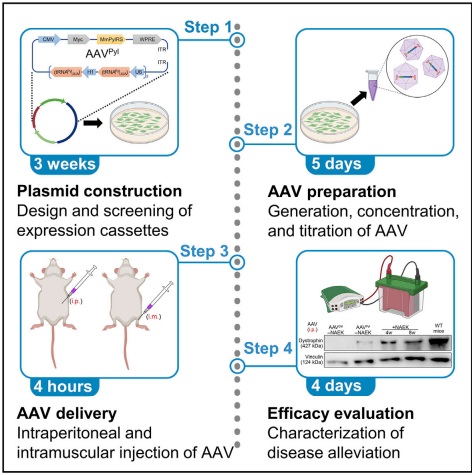
治疗研究离不开对于模型动物和递送方式的探讨。2023年3月17日,夏青团队受邀在Cell子刊STAR Protocols杂志在线发表了题为“Adeno-associated viral delivery of engineered tRNA-enzyme pairs into nonsense mutation mouse models”的实验方案文章(Protocol),旨在建立动物水平的实验流程和评价体系,推进这种新型疗法的临床转化研究。该文详细介绍了如何筛选和优化 tRNA-酶正交对以实现有效的 UAA引入,利用腺相关病毒(AAV)载体进行腹腔和肌肉注射的两种递送方式,并从恢复蛋白表达量、受累组织性状和动物行为学等多个维度详细评价最终的治疗效果。该工作进一步揭示此疗法的核心是制备新型生物正交的蛋白质翻译机器来通读无义突变位点并恢复功能性蛋白质的表达,从蛋白质编辑的角度来治疗遗传性疾病。

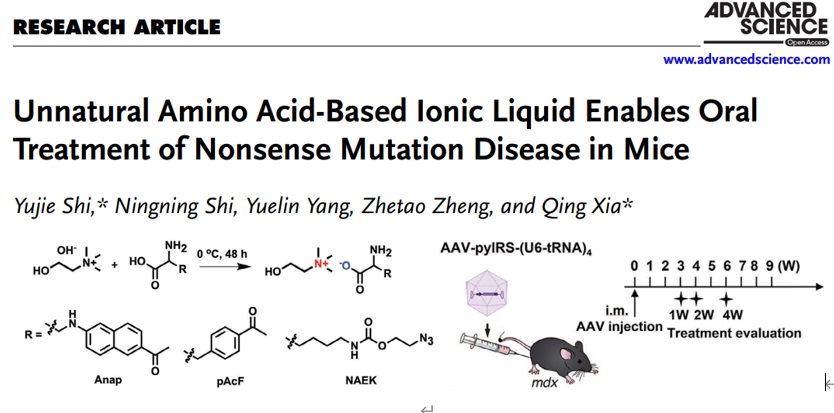
在此基础之上,夏青团队不断地更新迭代基因密码子扩展系统的递送方式。在实现tRNA-酶正交对的筛选和优化后,团队发现较低的UAA插入效率依旧限制了基因密码子扩展技术的转化治疗应用,原因在于UAA在血清中代谢迅速,难以在靶器官或靶组织中有效富集,从而导致现有策略中非天然氨基酸的体内利用率极低,限制了此方法的治疗潜力。为攻克该项难题,夏青团队首次将非天然氨基酸转化为离子液体,采用一种简单而经济的策略来优化UAA底物形式,实现无创而高效的口服给药,显著提高了UAA在体内的暴露及其在靶细胞中的利用率,并观察到更为快速、稳定和持久的蛋白恢复表达。该研究以“Unnatural amino acid-based ionic liquid enables oral treatment of nonsense mutation disease in mice”为题发表于Advanced Science期刊。
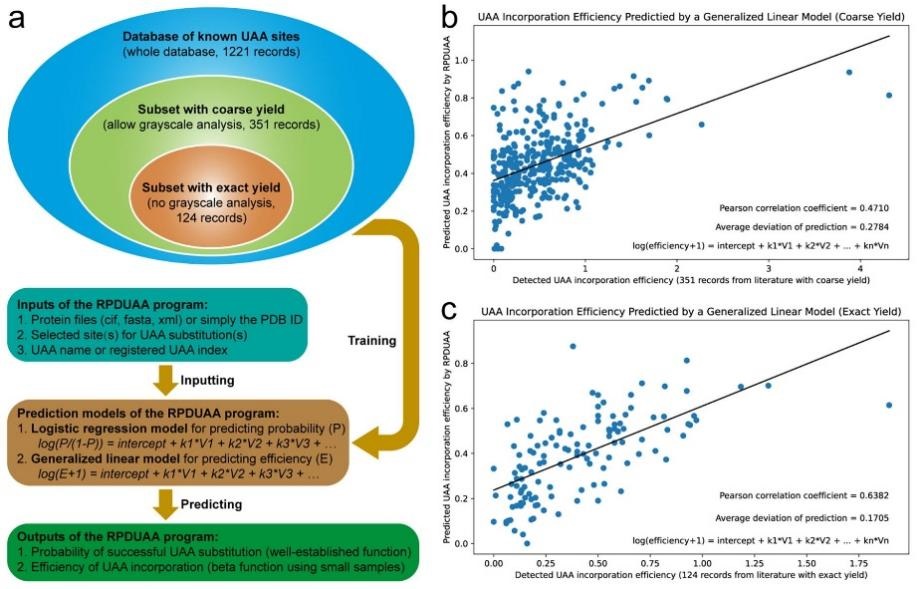
为了实现对任意非天然氨基酸插入任意蛋白上任意位点的成功概率和效率的预测,夏青团队开发了一款 AI 辅助非天然氨基酸插入蛋白质的软件。2022 年 9 月 8 日,相关论文以《机器学习辅助非天然氨基酸插入蛋白质的合理设计》(Rational incorporation of any unnatural amino acid into proteins by machine learning on existing experimental proofs)为题在Computational and Structural Biotechnology Journal上发表。
这项研究将 AI 应用于含非天然氨基酸蛋白的合理设计和虚拟筛选中,整理规范了此前非天然氨基酸 1221 个插入记录作为已知位点数据库,并对该库进行机器学习,把相关模型和算法编写成软件(Rational Protein Design with Unnatural Amino Acids,RPDUAA)。在研究病毒蛋白、无义突变致病的单基因蛋白或进行蛋白质修饰时,该软件能提供准确度在 80% 以上的非天然氨基酸插入位点的预测。该研究首次实现非天然氨基酸替换天然氨基酸的深度学习和精准预测,有利于实现无义突变位点的选择性,助推开启含有非天然氨基酸的新型蛋白质药物设计。
以在体应用基因密码子扩展技术的经验为基础,结合课题组关于抗病毒机制的研究积累,团队于2022 年 7 月 1 日发表“Sterilization of Drug-Resistant Influenza Virus through Genetic Interference Inspired by Unnatural Amino Acid-Engineered Particles”研究论文在Advanced Therapeutics期刊上。通过密码子扩展技术实现工程化病毒颗粒的高效生产;这种非天然氨基酸工程化的病毒颗粒,因病毒基因组中含有无义密码子,其复制增殖具有高度可控性。工程化病毒颗粒兼具预防型疫苗和治疗性药物的双重作用,通过病毒自身的重排规律,交换含有无义密码子的基因片段,能够高效且广谱地中和现行的流感病毒株和耐药流感病毒株,并不诱导产生新的耐药性。
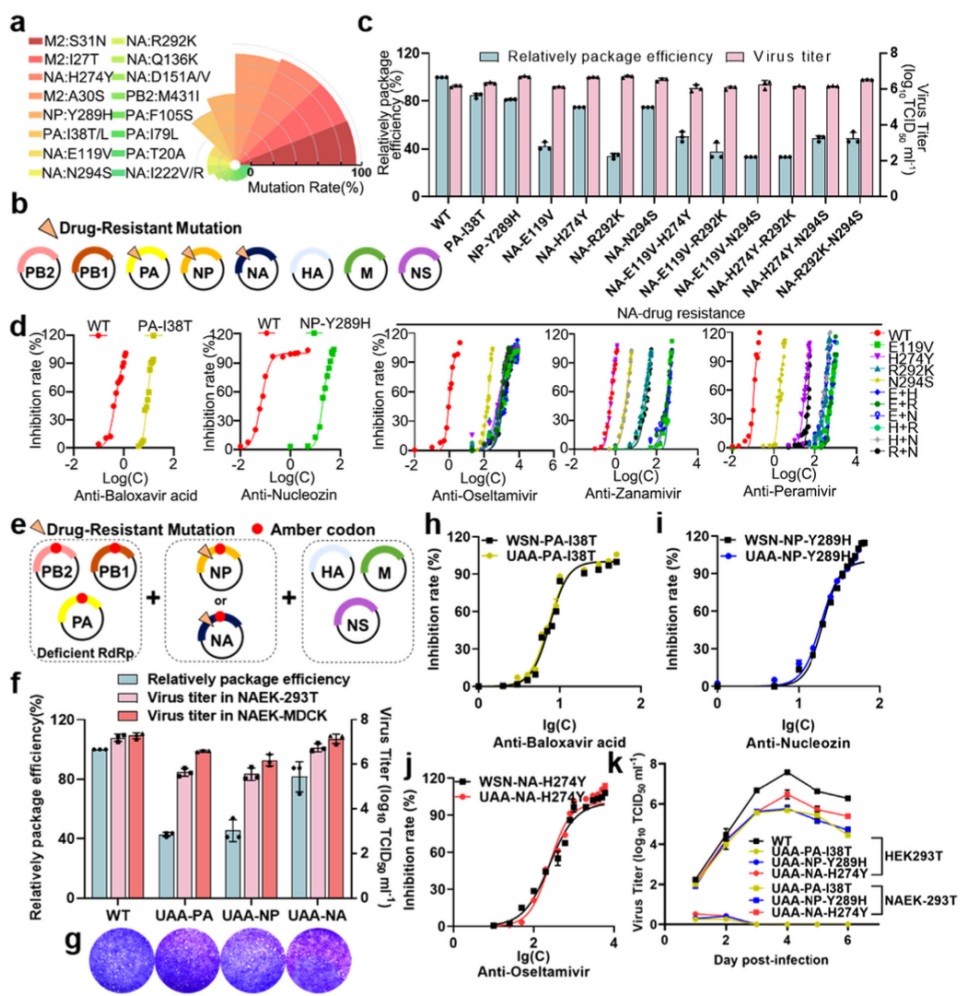
更进一步,团队实现了基因密码子扩展技术在单链RNA病毒的疫苗研究,于2023年5月30号在Vaccines期刊上发表题为“Attenuating RNA viruses with expanded genetic codes to evoke adjustable immune response in PylRS-tRNAPyl CUA transgenic mice”的研究文章,通过平行筛选得到单链RNA病毒基因组改造的最佳位点,并引入无义突变,在体外和体内利用基因密码子扩展技术实现对该改造病毒复制与增殖的人为控制,从而在转基因动物体内诱导强度可人为调节的免疫反应,其产生的抗体水平可在市售疫苗的0.5倍~2.5倍之间微调。该工作首次提出了“智能疫苗”的新理念,旨在安全可控的前提下可控制病毒疫苗的在体效力。2023年10月获批中国专利授权。

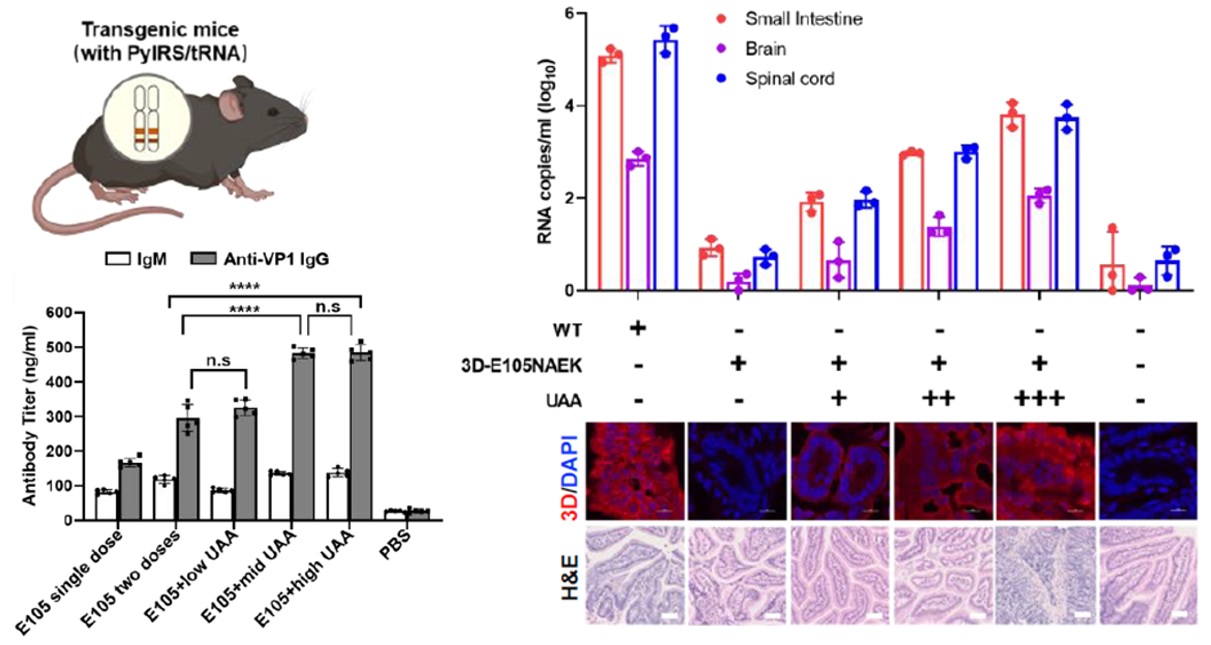
综上,团队在为Springer Nature旗下期刊Methods in Molecular Biology撰写的实验方法文章“Noncanonical Amino Acid Incorporation in Mice”中,以非基因密码子扩展系统转基因小鼠为工具,探讨了非天然氨基酸系统的治疗与工程应用,并深度总结了扩展此系统在体应用的前沿进展和潜在挑战,指出该技术从翻译水平恢复突变基因的蛋白表达,具有不同于基因编辑和基因治疗的独特治疗潜力。

【夏青教授简介】

夏青,北京大学药学院长聘教授,医学博士,博士生导师。研究方向为前沿生物技术与再生医学,主要从事工程细胞和类人器官中的蛋白质翻译机器与元件的系统改造,实现工程细胞与干细胞内的碱基、氨基酸和聚糖分子的精确替换。针对单基因病无义突变类型的治疗,制备新型蛋白质翻译机器,提高基因解码效率,控制蛋白翻译速度。这些应用会改变对单基因病点突变的理解,为治疗带来了新的理念。